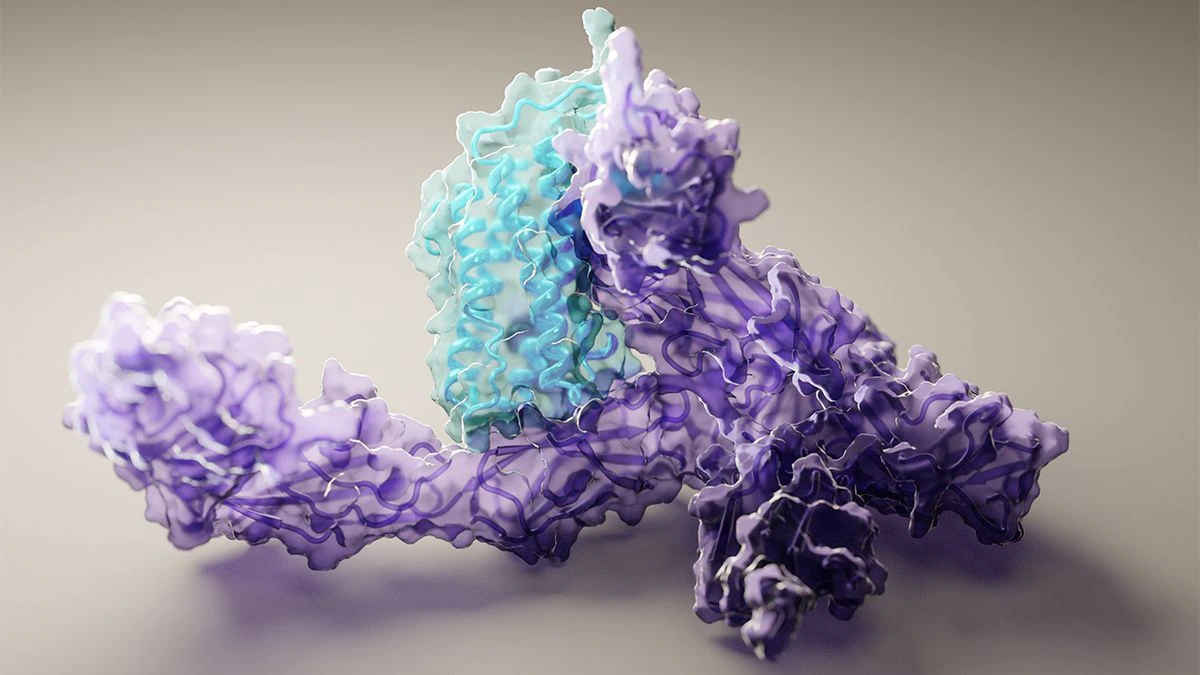
A veces es fácil llenarse la boca con expresiones como “santo grial”, “paso de gigante” o “descubrimiento revolucionario” al hablar de un logro científico. Pero cuando se trata de revelar la forma de las proteínas con la sencillez de quien busca algo en Google, esas palabras encajan como un guante. Este año se ha confirmado que la inteligencia artificial es capaz de adivinar la estructura de estos motores básicos de la vida, las proteínas, una de las claves fundamentales de la biología. Y es un logro por partida doble: “Primero, porque resuelve un problema científico que ha estado en la lista de tareas pendientes durante 50 años. En segundo lugar, es una técnica revolucionaria que acelerará enormemente el descubrimiento científico. Habría sido el Descubrimiento del Año por cualquiera de los dos motivos”, asegura Holden Thorp, director de la influyente revista científica Science, que concede el reconocimiento a este éxito científico. El año pasado aplaudieron el desarrollo de las vacunas contra la covid.
Este logro lo han realizado dos grupos distintos: la compañía DeepMind y el equipo de David Baker, bioquímico computacional de la Universidad de Washington. Un triunfo que hace apenas quince años parecía imposible de lograr, como recuerda el propio Thorp: “Cuando dejé el laboratorio en 2006, pensé que el problema del plegamiento de proteínas nunca se resolvería”. Muchos especialistas de ese campo creían que, de lograrse, no lo verían en vida. Una persona ajena al mundo de la biomedicina puede sorprenderse por la importancia que se le da a esta cuestión tan técnica, la de la forma en que se pliegan las proteínas; no parece un anhelo popular como la cura del cáncer o la vacuna de la malaria. Pero es que las proteínas, los operadores esenciales de casi todas las funciones biológicas, atraviesan esos y la mayoría de los problemas biomédicos imaginables. Incluida la covid. Una proteína es la espícula del coronavirus, los penachos que lo rodean y que le permiten conquistar nuestras células para enfermarnos. Conocer su estructura permite diseñar estrategias para atacar esa ganzúa con la que nos invade.
En 2006, pensaba que el problema del plegamiento de proteínas nunca se resolvería
Holden Thorp, director de ‘Science’
Casi todo el mundo tiene en mente la metáfora de que el ADN contiene las instrucciones de la vida. Aunque son las proteínas quienes la ponen a funcionar: realizan esas tareas, como activar el movimiento de los músculos, determinar el estado de los tejidos, transportar sustancias por el organismo o defenderlo de las agresiones. Las instrucciones del ADN por sí solas, lógicamente, no hacen nada. Ese manual nada más pone en marcha a las materias primas, los aminoácidos, sobre la cadena de montaje. Se disponen en fila en la cinta para que los operarios (los ribosomas) construyan esas máquinas que son las proteínas. Pliegan esa fila de aminoácidos para darle su complejísima forma a las proteínas, una estructura que determina su función: no es lo mismo construir una cafetera que un motor de coche.
La inteligencia artificial es capaz de saber si la cafetera será italiana, hexagonal y con asa de plástico, con solo ver los tornillos y las placas de latón dispuestas en fila bidimensional. Las proteínas se construyen combinando únicamente 20 aminoácidos en innumerables distribuciones y longitudes. Por eso, descifrar la disposición final en tres dimensiones es un trabajo arduo y costoso, que muchos laboratorios ni siquiera pueden plantearse. Descifrar esas estructuras en condiciones normales sería un esfuerzo que costaría miles de millones de años; incluso las proteínas de tamaño modesto podrían tomar un número astronómico de aspectos posibles. Con este logro de la computación, se consigue en un rato.
A mediados de julio, Baker y sus colegas publicaron en Science que su programa de inteligencia artificial RoseTTAFold había resuelto las estructuras de cientos de proteínas, todas importantes para la creación de medicamentos. A la vez y de manera independiente, los científicos de DeepMind difundieron en Nature su propio logro: su máquina, AlphaFold, había dibujado 350.000 proteínas que se encuentran en el cuerpo humano, el 44% de todas las proteínas humanas conocidas. Además, esperan que su base de datos crezca a 130 millones de proteínas pertenecientes a todas las especies, más de la mitad del número total que se cree que existe. Ambos equipos han puesto esas estructuras a disposición de todos los laboratorios del mundo, y también las herramientas para lograr otras nuevas.
No podía ser de otra manera, como expresó en su día Edith Heard, directora general del Laboratorio Europeo de Biología Molecular, de cuyos recursos se aprovechó DeepMind y con quien ahora comparte sus logros: “AlphaFold ha sido entrenado utilizando datos de recursos públicos creados por la comunidad científica, así que tiene sentido que sus predicciones sean públicas”. La investigadora lo mostraba como “una auténtica revolución para las ciencias de la vida, como lo fue la genómica hace décadas”.
Es una auténtica revolución para las ciencias de la vida, como lo fue la genómica hace décadas
Edith Heard, directora general del Laboratorio Europeo de Biología Molecular
Se abre la puerta a innumerables hallazgos en biomedicina, tratamientos y soluciones biológicas. “Todas las áreas de la biología molecular y computacional van a transformarse”, asegura Baker en Science. Las opciones son apasionantes: casi todos los medicamentos funcionan por asociarse a un punto específico de una proteína, por lo que conocer en detalle la diana multiplica la capacidad de acertar. Además, son muchas las enfermedades, desde el alzhéimer y el párkinson a la diabetes o la fibrosis quística, que se generan tras la acumulación de proteínas mal plegadas, por lo que ahora se podrían abordar con mejor pronóstico. Y no solo eso: al saber cómo se doblará una cadena bidimensional de aminoácidos, aparece la posibilidad de diseñar proteínas artificiales: que protejan de los virus, como ya se está ensayando, pero también para descomponer materiales contaminantes.
“No sabía qué era una proteína cuando empecé [en DeepMind]”, reconoce John Jumper, responsable de este hallazgo para la compañía, hermana de Google bajo el paraguas de Alphabet. Aplicar la inteligencia artificial a un problema científico fue “realmente mágico”, asegura Jumper en declaraciones a Nature, la prestigiosa revista científica que le ha incluido entre los diez científicos más relevantes de 2021. Su logro, publicado en esa revista en julio, contaba con dos españoles entre los firmantes: Oriol Vinyals y Bernardino Romera.
El siguiente paso será predecir cuáles de esas proteínas descritas trabajan juntas y cómo interactúan, porque en muchas ocasiones no funcionan solas. La inteligencia artificial ya está afanada en ello tras demostrar esta vez que sí es capaz de cumplir los ambiciosos éxitos que llevaba años prometiendo. Y no sería raro que esto termine en Estocolmo celebrando un Nobel: la importancia de las proteínas se descubre de un vistazo al ver los numerosos premios concedidos en Química o Medicina a descubrimientos ligados a la estructura de estos motores de la vida. Los primeros en describir proteínas, como Frederick Sanger, John Kendrew y Max Perutz recibieron sus galardones, hace seis décadas. Ahora describen miles de un plumazo.
Puedes escribir a javier@esmateria.com y seguir a MATERIA en Facebook, Twitter e Instagram, o apuntarte aquí para recibir nuestro boletín semanal.